能高效分析細胞分裂4D圖像數據的超強運算工具
由香港城市大學(香港城大)科學家共同領導的聯合研究團隊,開發出一種可以重建和以三維(3D)圖像呈現細胞形狀隨著時間而變化的新型運算工具,令分析過程從需要人手操作數百小時,加快至只需用電腦數小時便可完成。此運算工具顛覆了生物學家分析圖像數據的方式,有助推進細胞和發育生物學、如癌細胞生長的進一步研究。
該跨學科研究由香港城大電機工程學系(EE)電腦工程學講座教授兼黃俊康教授(數據工程)嚴洪教授,聯同香港浸會大學以及北京大學的生物學家共同領導。研究成果已於科學期刊《自然通訊》(Nature Communications)上發表,題為〈Establishment of a morphological atlas of the Caenorhabditis elegans embryo using deep-learning-based 4D segmentation〉。
團隊開發出來的運算工具名為「CShaper」,嚴教授形容說︰「它是一個強大的運算工具,能於單細胞層面上有系統地分割細胞圖像和分析,對有關細胞分裂和細胞與基因功能的研究而言,非常有用。」

分析大量細胞分裂數據的瓶頸
生物學家一直努力研究動物怎樣經由無數次的細胞分裂,從單一細胞、即受精卵,演化成各個器官和整個身體。他們尤其渴望揭示基因的功能,例如哪些特定的基因於細胞分裂過程中形成各種不同器官,或是甚麼導致異常的細胞分裂而產生腫瘤。
採用基因敲除技術(gene knockout technique)是其中一個找出答案的方法。研究人員會首先在全部基因齊全的情況下取得細胞圖像和譜系樹(lineage tree),然後從DNA序列中「敲除」(剔除)一個基因,再比較兩組譜系樹以分析細胞的變化,並推斷出基因功能;然後重複做法,逐一「敲除」其他基因,再逐一比較。
合作參與這個研究的生物學家團隊選用了秀麗隱桿線蟲(Caenorhabditis elegans, C. elegans)的胚胎作為研究對象,並得出數以兆計字節(terabytes)的海量數據,供嚴教授的團隊進行運算分析。秀麗隱桿線蟲是一種蠕蟲,具有許多與人類相同的重要生物學特性,因而為研究人類腫瘤的生長過程提供參考價值。
嚴教授說:「秀麗隱桿線蟲約有20,000個基因,這意味著若每次只敲除一個基因的話,將需要進行近20,000次實驗,產生大量的數據,所以必需運用自動化圖像分析系統,也因此驅使我們開發出一個更高效的相關系統。」
自動分割細胞圖像的突破
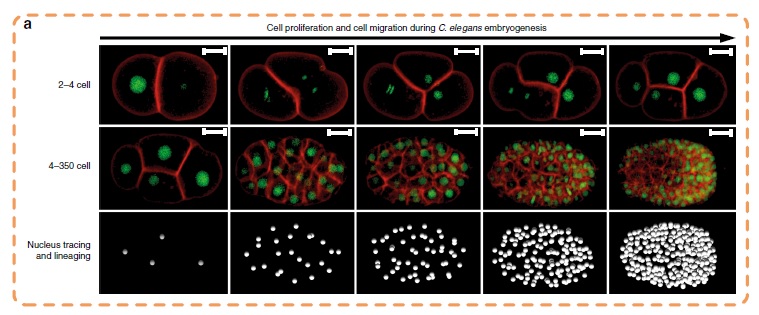
研究人員一般利用激光束掃描,而取得細胞圖像。不過,現有的圖像分析系統只能擷取到細胞核圖像,但是細胞膜的圖像質量差劣,難以重建細胞形狀;而且,現時缺乏可靠的算法,能夠把記錄細胞分裂過程的時序(time-lapsed)3D圖像(即4D圖像)分割。所謂圖像分割(image segmentation)是指將視覺訊號劃分為多個部分以簡化圖像分析,是電腦視覺技術的一個關鍵過程,但研究人員往往動輒需要花上數以百計的小時,以手動方式標記眾多細胞圖像。
CShaper的突破之處,在於它能擷取細胞膜形狀、構建出細胞3D形狀,更重要的是,它能夠自動分割細胞圖像。嚴教授課題組的博士生、論文第一作者之一的曹劍鋒說:「生物學家透過使用CShaper,可以在數小時內解讀這些圖像。CShaper可以表徵出細胞的形狀和表面結構,並提供細胞在不同時間點的3D圖像。」
這個突破可以歸功於由團隊開發、基於深度學習的模型DMapNet,在CShaper系統裡所發揮的關鍵作用。嚴教授解釋說:「通過學習『捕獲』圖像像素之間的多重離散距離(multiple discrete distances),DMapNet在擷取細胞膜輪廓時會一併考慮形狀,而非僅僅考慮圖像光暗強度特徵。因此,CShaper辨認細胞的準確度高達95.95%,遠勝於其他方法。」
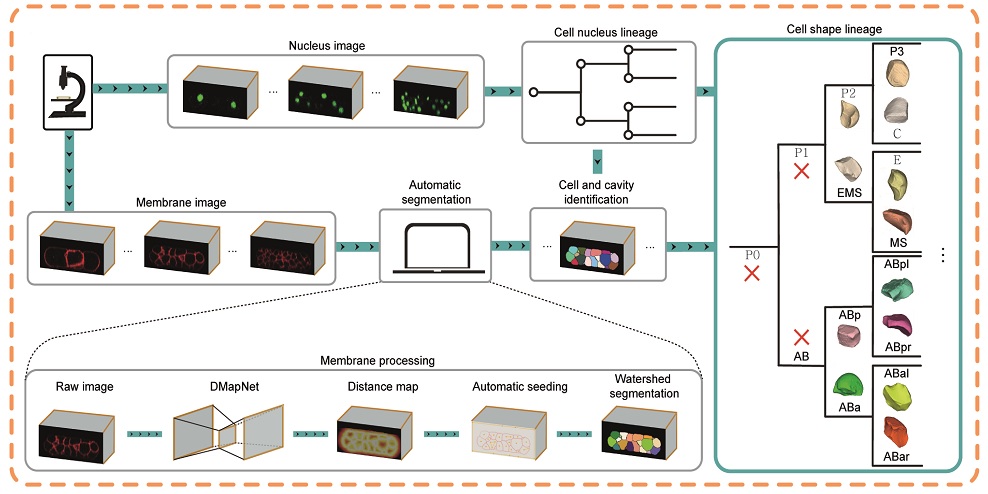
團隊運用CShaper,構建出秀麗隱桿線蟲的胚胎由4到350細胞期的3D細胞形態時序圖譜,內容涵蓋細胞形狀、體積、表面積、細胞遷移、細胞核位置以及細胞之間的接觸,並識別出細胞身份。
推進腫瘤生長的研究
曹劍鋒說:「據我們所知,CShaper是首個能在單細胞層面上,有系統地對秀麗隱桿線蟲的胚胎進行圖像分割和分析的運算系統。」嚴教授補充說:「透過與生物學家的緊密合作,很高興我們開發出一種可以自動分析大量細胞圖像數據的電腦工具。我們相信,這套運算系統可以促進細胞生物學和細胞發育學的進一步研究,特別是了解癌細胞的起源和生長。」
團隊亦以植物的組織細胞測試CShaper,得出令人鼓舞的结果,他們相信此運算工具可以應用於其他生物學研究。
嚴教授與來自北京大學的湯超教授、以及來自香港浸會大學的趙中應教授是論文的共同通訊作者。論文的共同第一作者分別是香港城大EE學系的博士生曹劍鋒、來自北京大學的關國業,與香港浸會大學的何穎詩 。

這項研究獲得美國國家衛生研究院、香港研究資助局、中國科技部與國家自然科學基金會的資助。
DOI number: 10.1038/s41467-020-19863-x
